Cada novo fármaco pode demorar uma dezena de anos desde os primeiros estudos até chegar ao balcão das farmácias. Pelo caminho ficam várias moléculas, tentativas que não resultaram, ensaios clínicos que não correram como esperado. E um dos motivos para “arruinar” um potencial bom medicamento é a transformação que sofre no nosso fígado. O trabalho liderado por uma equipa portuguesa do Laboratório de Cristalografia Molecular da Universidade Nova de Lisboa, publicado esta segunda-feira na revista científica Nature Chemical Biology, promete facilitar a vida a quem estuda o desenvolvimento de novos fármacos.
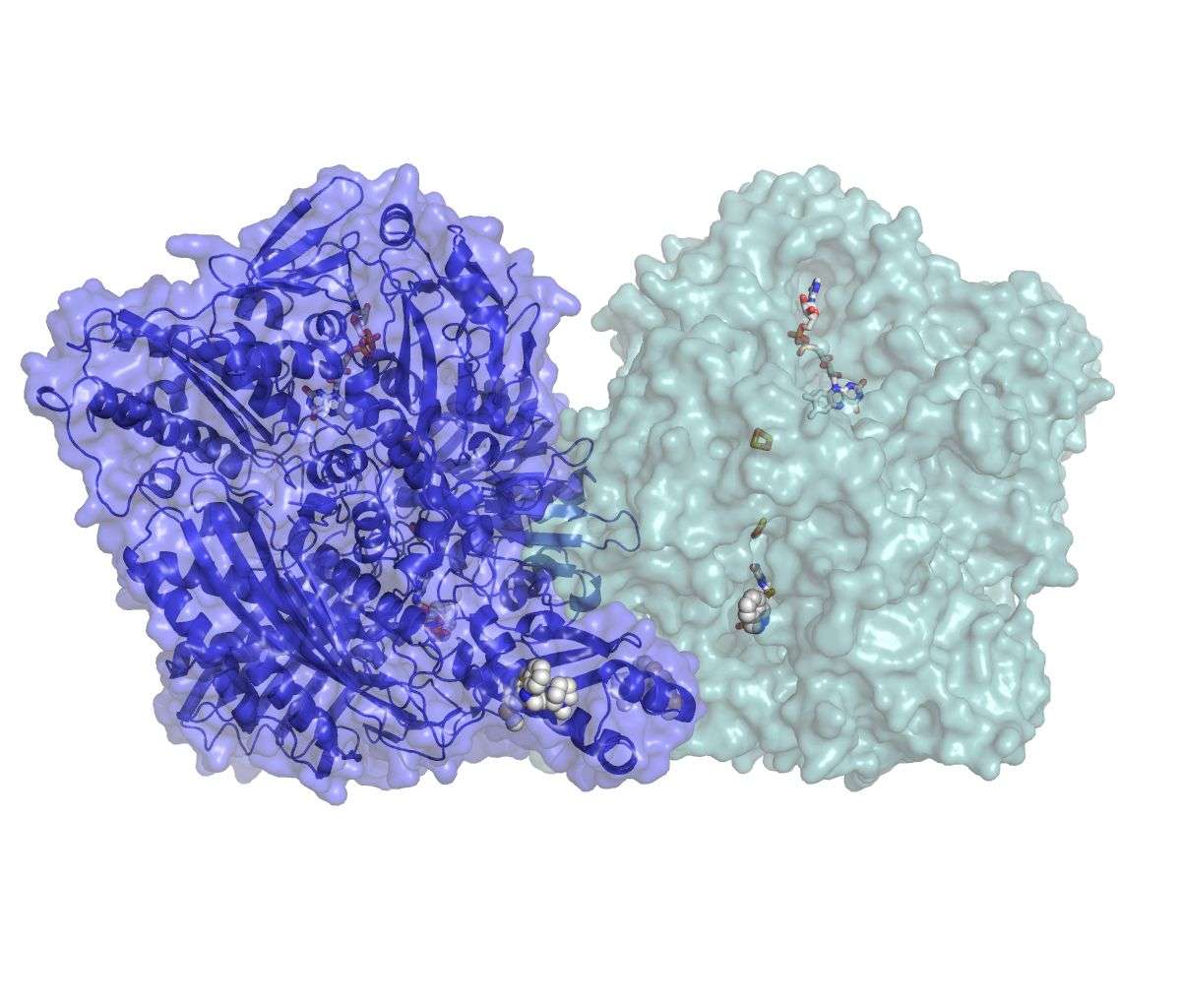
O que a equipa da unidade de investigação Ucibio-Requimte, em conjunto com os investigadores do Instituto para a Bioquímica e Biologia da Universidade de Postdam (Alemanha), fez foi determinar a estrutura tridimensional da proteína aldeído oxidase humana (AOX) – uma proteína do fígado responsável por metabolizar (degradar e transformar noutras moléculas) os fármacos. Perceber como é que a proteína se liga às outras moléculas pode ajudar a perceber como ela funciona.
“Neste trabalho conseguimos cristalizar e determinar a estrutura 3D da AOX humana, o que permitiu compreender a respetiva ação catalítica e modo de inibição”, diz em comunicado de imprensa Maria João Romão, líder do grupo de de Cristalografia Molecular. “Esta informação irá contribuir para melhorar o processo de desenho de fármacos, permitindo que a farmacêuticas possam desenvolver métodos in silico de previsão do metabolismo pela AOX.”
Os ensaios pré-clínicos preveem que os medicamentos sejam testados em ratos de laboratório ou outras cobaias, mas enquanto os roedores têm quatro formas diferentes desta proteína, os humanos têm apenas uma. Logo não é possível extrapolar o que acontece num rato para o que poderá acontecer num humano. Portanto, um novo fármaco pode até ter passado todos os ensaios e mostrar-se seguro com humanos, mas ser pouco ou nada eficaz para tratar uma doença porque o fígado o destrói de uma forma imprevista antes de cumprir a sua função. Conhecer a estrutura da enzima poderá ajudar a ultrapassar esta limitação. Quanto menos tempo e menos becos sem saída tiver o desenvolvimento de um novo fármaco, menos custos terá.
Podemos ter um conjunto de ripas de madeiras, que identificamos como potencialmente pertencendo a uma peça de mobiliário, mas só quando montamos as várias partes é que percebemos que é uma cadeira e que serve para sentar. O mesmo acontece com as proteínas. Podem ser conhecidas todas as peças que as compõe – os aminoácidos -, mas só quando estão organizados numa estrutura tridimensional é possível perceber como é que a proteína funciona.
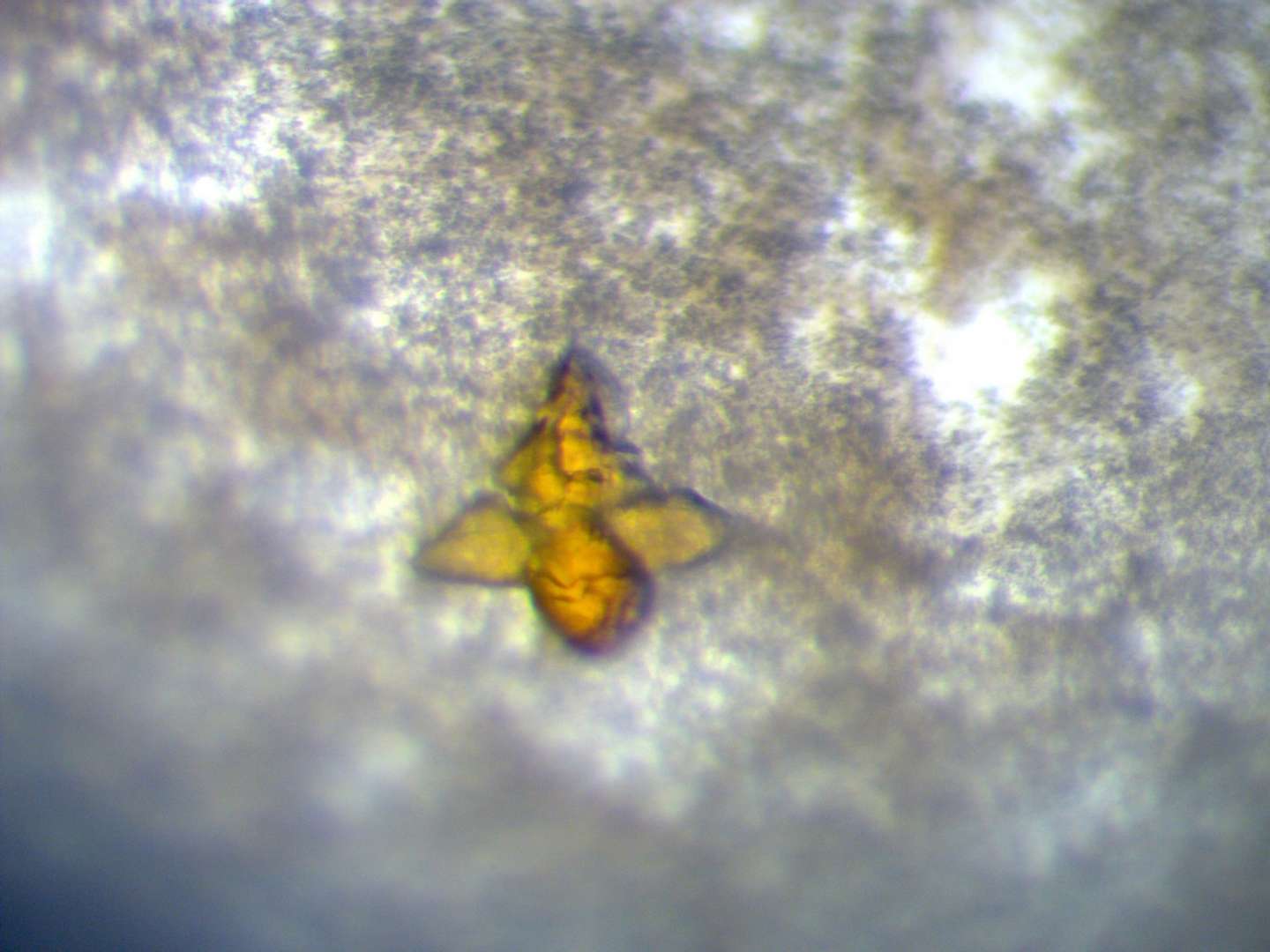
A aldeído oxidase humana tem mais de 1.330 aminoácidos, que no seu conjunto representam mais de 10 mil átomos, e foram precisos oito anos para finalmente conseguir determinar a estrutura tridimensional da enzima humana. Nos últimos quatro ano a equipa criou mais de 800 cristais – estruturas tridimensionais que potencialmente poderiam ser a estrutura real da proteína – e viajou mais de 40 vezes aos laboratórios de radiação de sincrotrão para conseguir fazer os raios-X necessário para encontrar a proteína “certa”.
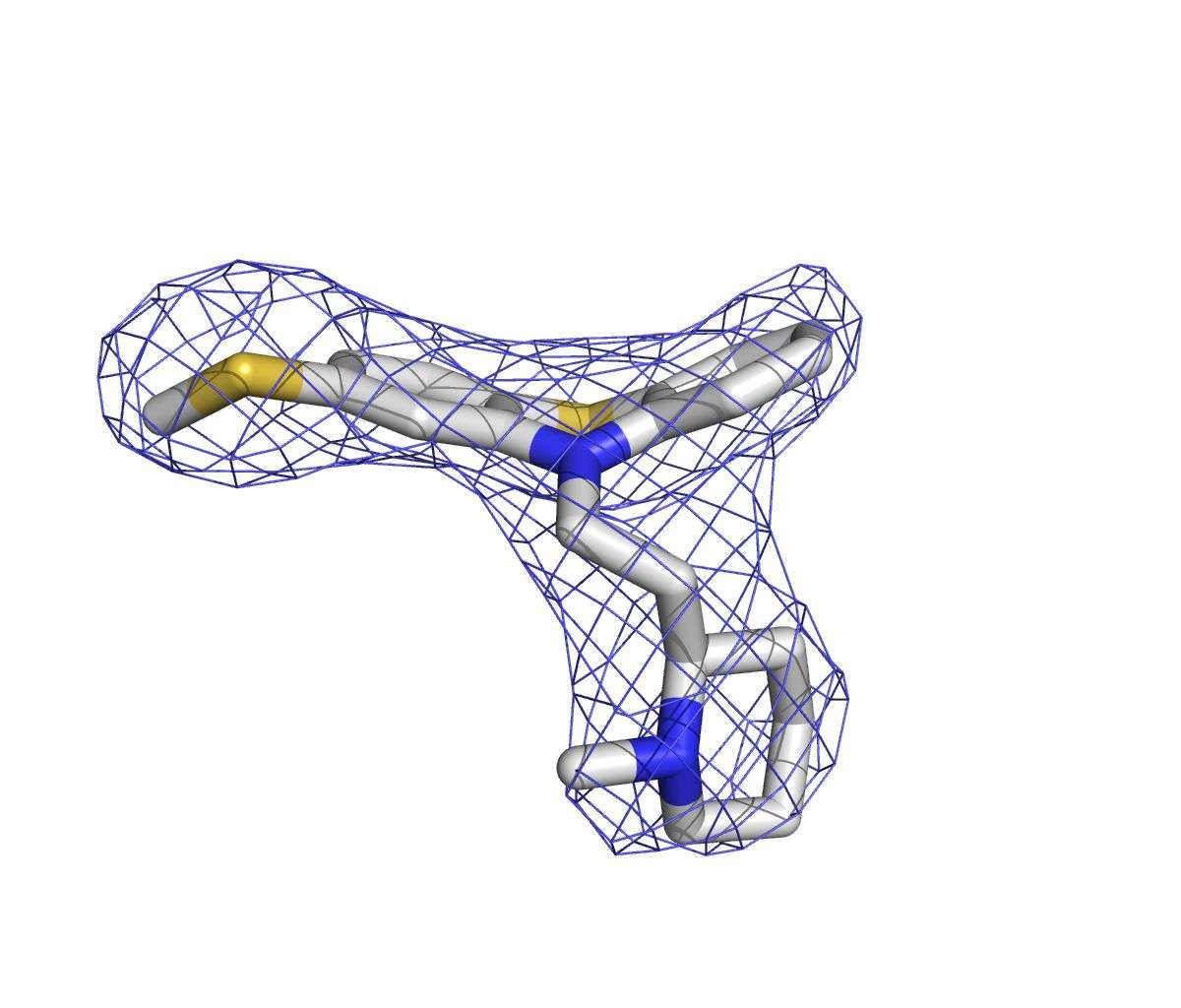
Como resultado destas experiências, as autoras puderam determinar a estrutura da enzima nativa a solo assim como ligada a um antipsicótico (tioridazina), refere o comunicado de imprensa. “Os dados que obtivemos revelaram um novo local de ligação que pode ter um carácter geral para outros fármacos análogos”, explica Teresa Santos Silva, coautora do artigo. Desenhar fármacos resistentes ao metabolismo da AOX poderia ajudar a reduzir a taxa de insucesso dos ensaios clínicos.